根据《细胞报告》上发表的一项最新研究,西北大学和伊利诺伊大学香槟分校(UIUC)的科学家使用一种新颖的综合计算技术,可以使用表观基因组数据集在分子水平上对疾病状况进行分类。
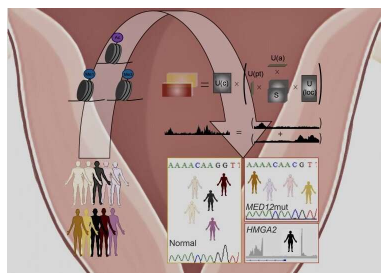
由西北和UIUC的作者合作开发的新方法称为DeCET(表观基因组张量的分解和分类)。它分析复杂的异构数据,以识别组织类型,疾病亚型和细胞类型变化或细胞分化之间的表观基因组差异,转化研究副主席,安娜·拉普汉姆妇产科学教授Debabrata Chakravarti博士解释说。并是该研究的主要作者。
Chakravarti教授说,表观基因组是对组蛋白和基因组进行修饰的所有修饰的集合,这些基因组可以改变基因的表达,因此具有指明细胞身份的基本说明,因此表观基因组测定可以为多种疾病提供可靠的诊断标记。罗伯特·H·卢里综合癌症中心药理学和共享资源助理主任。例如,DNA甲基化的模式可以对原发性和继发性中枢神经系统肿瘤进行分类,并可以确定癌症起源的细胞类型。
UIUC的物理学创始人教授Jun Song博士和西北大学内分泌学医学副教授Grant Barish博士是该研究的共同资深作者,Priyanka Saini博士是该研究的共同作者。 Northwestern的研究者和UIUC的Jacob Leistico是研究的共同第一作者。Leistico领导了算法开发,而Saini领导了实验工作,在两个机构之间建立了一个协同团队。
由基础科学家,临床医生和物理学家组成的合作小组对子宫肌瘤或平滑肌瘤进行了初步研究,子宫肌瘤是子宫平滑肌细胞的非癌性肿瘤,治疗选择有限,多达70%的女性会发展为子宫肌瘤。
科学家首先对来自患者的25例正常子宫和25例匹配的肌瘤样本进行了表观基因组分析。然后,该团队使用DeCET分析数据并确定关键的表观基因组特征,这些特征可以清楚地区分正常的子宫肌层组织与平滑肌瘤的疾病状态和疾病亚型。
研究人员接下来将DeCET应用于“未知”人类样本以预测组织状态。该方法使用表观基因组特征准确区分正常和疾病类型以及亚型。
最后,为了证明DeCET的普遍适用性,研究小组使用了公开的癌症表观基因组数据集扩展了他们的分析范围,并成功地将其他疾病(例如乳腺癌和前列腺癌)进行了分类。
“我们的研究克服了当前分析方法的缺陷,例如荟萃分析参数的任意选择和样本间变异性,” Saini和Leistico解释说。
据Barish称,DeCET在分层不同的组织类型,疾病亚型和细胞分化状态方面也优于现有工具。
Chakravarti说:“表观基因组学分析是通向个性化医学的途径。”
监督计算算法开发的Song补充说:“我们希望我们的研究将有助于更好地理解和识别疾病状态,从而为将来的治疗,诊断和预后策略提供便利。”
标签: 生物信息计算工具
郑重声明:本文版权归原作者所有,转载文章仅为传播更多信息之目的,如作者信息标记有误,请第一时间联系我们修改或删除,多谢。